![next]()
![up]()
![previous]()
![contents]()
Next: Diverse metoder Up: Metoder Previous: Kloning i Saccharomyces cerevisiae
Subsections
Med DNA-sekvensering forstår vi bestemmelse av baserekkefølgen i et
DNA-molekyl. Sangers kjedetermineringsmetode ligger til grunn for de fleste
sekvenseringsteknikker som brukes i dag. Grunnlaget for metoden ligger i
dideoksynukleotiders terminerende effekt på ulike DNA polymerasers aktivitet.
Templat (dobbelt- eller enkelttrådig DNA), primer, dNTP og en DNA polymerase
uten exonuklease-aktivitet blandes med en liten andel av et dideoksynukleotid.
Polymeriseringen vil terminere vilkårlig ved inkorporering av dideoksynukleotid
istedet for det tilsvarende deoksynukleotid.
Separate reaksjoner for hvert av de fire nukleotidene vil gi distinkte
blandinger av terminerte DNA-fragmenter som kan skilles ved elektroforetiske
teknikker. Fragmentene kan detekteres ved ulike teknikker som radioaktiv eller
fluoriserende merking. Bruk av fluoriserende primer eller dNTP gir mulighet for
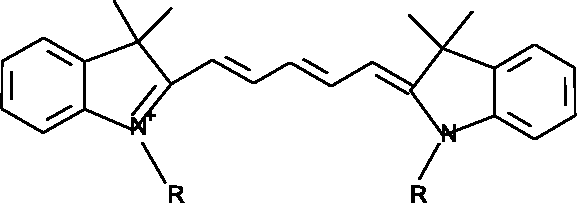
automatisering av sekvenseringen. En mye brukt fluoriserende kjemisk gruppe er
Cy5, som er et indokarbocyanin (se figur 3.5)
Figure 3.5: Generell molekylstruktur for
indokarbocyaniner
|
Varmestabile polymeraser og PCR-teknologi har gjort sekvensering til en meget
følsom teknikk med mindre strenge krav til mengde templat.
Sekvenseringsreaksjonene kjøres i sykler av denaturering, annealing og
elongering, som gir lineær amplifisering av terminerte fragmenter. Denne
varianten av sekvensering kalles ofte ``cycle sequencing''
Prosedyre -
Syklussekvensering med radioaktiv inkorporering
Prosedyren nedenfor er basert på protokollen for PERKIN
ELMER`S AmpliCycle kit (#N808-0175), og gjør på bruk av [  P]-dCTP eller -dATP.
P]-dCTP eller -dATP.
- 1.
- Tilsett for hver prøve som skal sekvenseres 2
 l av hver av termineringsløsningene G, A, T og C til
hvert sitt PCR-rør
l av hver av termineringsløsningene G, A, T og C til
hvert sitt PCR-rør
- 2.
- Lag en reaksjonsblanding, som vist i tabell 3.6,
for hver prøve.
- 3.
- Pipetter 6
 l
av reaksjonsblandingen til hvert av de 4 tilhørende PCR-rørene fra punkt 1.
l
av reaksjonsblandingen til hvert av de 4 tilhørende PCR-rørene fra punkt 1.
- 4.
- Tilsett 20
 l
mineralolje dersom PCR-maskinen (programmerbar varmeblokk) som skal benyttes
ikke har varme i lokket.
l
mineralolje dersom PCR-maskinen (programmerbar varmeblokk) som skal benyttes
ikke har varme i lokket.
- 5.
- Sett alle PCR-rørene i programmerbar varmeblokk og varm opp til 95
 C
i 1 minutt. Kjør deretter 25 sykluser av temperaturprogrammet vist i tabell 3.7
.
C
i 1 minutt. Kjør deretter 25 sykluser av temperaturprogrammet vist i tabell 3.7
.
Table 3.7: Temperaturprogram syklussekvensering
med radioaktivt merket primer
| Trinn |
Temperatur (  C) C) |
Tid (s) |
| 1 |
95 |
30 |
| 2 |
68 |
30 |
| 3 |
72 |
60 |
|
- 6.
- Etter endt reaksjon tilsettes 4
 l
stoppløsning til hvert rør. Bland ved å knipse forsiktig på røret. Spinn raskt
ned hvis nødvendig. Holdes kaldt.
l
stoppløsning til hvert rør. Bland ved å knipse forsiktig på røret. Spinn raskt
ned hvis nødvendig. Holdes kaldt.
- 1.
- Vask glassplatene og 2 spacere med destillert H2O og deretter
70% etanol.
- 2.
- Monter glassplatene mot hverandre med en spacer på hver side. Fest platene
sammen med tape (ikke klemmer).
- 3.
- Tilsett 12 ml Sequagel Complete til 48 ml Sequagel-6 (3.3.1.2.4)
og avgass. Dette gelvolumet passer til sekvenseringsapparat fra IBI.
- 4.
- Tilsett 400
 l
10% APS og bland godt. Sug gelløsningen opp i en sprøyte og før den forsiktig
inn mellom platene, som ligger horisontalt. Bank forsiktig ved væskefronten
for å unngå bobler.
l
10% APS og bland godt. Sug gelløsningen opp i en sprøyte og før den forsiktig
inn mellom platene, som ligger horisontalt. Bank forsiktig ved væskefronten
for å unngå bobler.
- 5.
- Sett i kam. Fest 2 klyper over glassplatene ved kammen. La gelen
polymerisere minst 1 time.
- 1.
- Fjern kammen forsiktig og monter gelen i elektroforeseapparatet.
- 2.
- Fyll 1xTBE i bufferkarene og spyl brønnene.
- 3.
- Preelektroforer gelen i ca
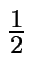 time ved 55W.
time ved 55W.
- 4.
- Denaturer prøvene 2 minutter ved 95
 C. Hold dem deretter på is.
C. Hold dem deretter på is.
- 5.
- Appliser 3
 l
av hver prøve. Noter appliseringsrekkefølge.
l
av hver prøve. Noter appliseringsrekkefølge.
- 6.
- Elektroforer ved 55W i 2-3 timer. Optimal elektroforesetid avhenger av
hvilket område man vil lese sekvens i.
- 7.
- Slå av spenningen og demonter gelen. La den avkjøles 5 minutter med is på
begge sider.
- 8.
- Overfør gelen til et 3MM papir (klippet ut slik at det passer til
eksponeringskassen) og skyll over med 4x 25ml 10% eddiksyre.
- 9.
- Plasser gelen med plastfolie over på geltørker. Tørk gelen ved vakuum og
65
 C
i ca 1 time.
C
i ca 1 time.
- 10.
- Plasser 3MM papiret med tørket gel over en røntgenfilm i
eksponeringskassett. Gelsiden legges mot filmens eksponerbare side.
Eksponeringstid avhenger av mengde templat og radioaktivitet. 3 døgns
eksponering har ofte vært brukbart til 25 fmol templat og 2,5
 Ci radioaktivitet.
Ci radioaktivitet.
Løsninger
Nedenfor er innholdet i de kommersielle løsningene levert med ``Cycle
sequencing kitet'' og gelløsningene gjengitt.
22,5  M
c7-dGTP
M
c7-dGTP
10  M
dATP, dCTP, dTTP
M
dATP, dCTP, dTTP
600  M
ddATP
M
ddATP
22,5  M
c7-dGTP
M
c7-dGTP
10  M
dATP, dCTP, dTTP
M
dATP, dCTP, dTTP
300  M
ddCTP
M
ddCTP
22,5  M
7-deaza-dGTP (c7-dGTP)
M
7-deaza-dGTP (c7-dGTP)
10  M
dATP, dCTP, dTTP
M
dATP, dCTP, dTTP
80  M
ddGTP
M
ddGTP
22,5  M
c7-dGTP
M
c7-dGTP
10  M
dATP, dCTP, dTTP
M
dATP, dCTP, dTTP
900  M
ddTTP
M
ddTTP
0,25 U/ l
AmpliTaq DNA polymerase CS
l
AmpliTaq DNA polymerase CS
500 mM Tris-HCl, pH 8,9
100 mM KCl
25 mM
MgCl2
0,25% (v/v) Tween 20
42,5 % Formamid
20 mM EDTA
0,05 % Bromfenolblått
0,02 % Xylene
Cyanol FF
5,7% (w/v) akrylamid
0,3% (w/v) methylen-bis-akrylamid
urea
(konsentrasjon ikke oppgitt, hemmeligholdt av produsenten)
0,1 M Tris-borat
- 2 mM EDTA buffer (pH 8,3)
Innholdet hemmeligholdes av produsenten.
Prosedyre - Syklussekvensering med
fluoriserende primer og automatisk sekvenator
Prosedyren som følger er basert på AMERSHAMs ``Thermo
Sequenase fluorescent labelled primer cycle sequencing kit'' (RPN 2436) og
protokoll for bruk av PHARMACIAs ALFExpress automatiske
sekvenator.
Bruk Cy5-merket primer med lengde på minst 18 basepar. Primeren fortynnes til
brukskonsentrasjon på 1-2  M.
Templatet kan være enkelttrådig eller dobbeltrådig DNA av høy kvalitet. Primer
bør være komplementær til et område i templatet minst 40 basepar fra starten av
den ønskede sekvensen.
M.
Templatet kan være enkelttrådig eller dobbeltrådig DNA av høy kvalitet. Primer
bør være komplementær til et område i templatet minst 40 basepar fra starten av
den ønskede sekvensen.
- 1.
- Tin følgende av løsningenen i kitet på is (se avsnitt 3.3.1.3.5):
A ,C, G, T og formamid loading buffer.
- 2.
- Fortynn 0,5-5
 g
av hvert templat til totalt 21
g
av hvert templat til totalt 21 l.
Templatmengder nær øvre grense gir best sekvens.
l.
Templatmengder nær øvre grense gir best sekvens.
- 3.
- Merk 4 PCR-rør for hvert templat og tilsett 2
 l av løsningene A, C, G og T til hvert sitt rør. Tilsett
deretter 1
l av løsningene A, C, G og T til hvert sitt rør. Tilsett
deretter 1  l
primer (1-2
l
primer (1-2 M)
til hvert rør.
M)
til hvert rør.
- 4.
- Tilsett 5
 l
templat-løsning til hvert av de 4 tilhørende PCR-rørene. Bland godt. Primer og
templat kan godt blandes på forhånd, for å forenkle pipetteringsarbeidet.
Tilsett litt mineralolje dersom PCR-maskinen som skal benyttes ikke har varme
i lokket.
l
templat-løsning til hvert av de 4 tilhørende PCR-rørene. Bland godt. Primer og
templat kan godt blandes på forhånd, for å forenkle pipetteringsarbeidet.
Tilsett litt mineralolje dersom PCR-maskinen som skal benyttes ikke har varme
i lokket.
- 5.
- Kjør termosyklus-reaksjoner i PCR-maskin. Denaturer først 5 minutter ved
95
 C.
Deretter kjøres 25 sykler som vist i tabell 3.8.
C.
Deretter kjøres 25 sykler som vist i tabell 3.8.
- 6.
- Når reaksjonen er ferdig, tilsett 3
 l
formamid loading buffer. Oppbevar prøvene på is eller ved 4
l
formamid loading buffer. Oppbevar prøvene på is eller ved 4  C.
C.
- 7.
- Før applisering på sekvenseringsgel denatureres prøvene 2 minutter ved 95
 C
C
Table 3.8: Temperaturprogram Amersham
syklussekvensering
| Tid (s) |
Temperatur (  C) C) |
Aktivitet |
| 30 |
95 |
Denaturering |
| 30 |
60 |
Annealing og elongering |
|
Nedenfor er fremgangsmåten ved støping av sekvensgel til ALFexpress
automatisk sekvenator beskrevet. Både Long Ranger akrylamid og vanlig akrylamid
(se punkt 3.3.1.3.5)
kan benyttes til støping av gel. Med Long Ranger gel og godt templat kan man på
det beste lese opp til 1000 basepar sekvens med metoden. Vanlig akrylamidgel gir
bedre oppløsning enn Long Ranger gel i området 0-300 basepar
- 1.
- Ta ut 50 ml 6% gel-løsning fra kjøleskap og la stå i romtemperatur til den
er temperert.
- 2.
- Vask begge glassplatene og to 0,5 mm tykke spacere nøye med destillert
vann og deretter 70% etanol.
- 3.
- Påfør et meget tynt lag med Bind-Silane (se punkt 3.3.1.3.5)
der hvor brønnene skal være. La tørke minst 20 minutter ved romtemperatur.
Bind-Silane binder gelen kovalent til glassplatene, slik at brønnene blir mer
robuste. Poler til slutt glassplatene med papir fuktet med destillert vann og
fjern eventuelle fiber og støvpartikler.
- 4.
- Monter platene med spacerne mellom. Sørg for at spacerne ligger helt ut
til kanten av platene. Fest den øvre glassplaten til den nedre med 4 klemmer
på hver side. Pass på at spacerne er fri for støv der hvor laser-strålen skal
gå. Sett i kam øverst slik at den er helt i flukt med høyre kant sett fra
forsiden av gelen.
- 5.
- Filtrer gelløsningen med sug slik at man i tillegg oppnår en viss
avgassing.
- 6.
- Tilsett 300
 l
10% ammoniumpersulfat-løsning og 30
l
10% ammoniumpersulfat-løsning og 30  l
TEMED til gelløsningen (Long Ranger). De tilsvarende volumene for vanlig
akrylamidgel er 250
l
TEMED til gelløsningen (Long Ranger). De tilsvarende volumene for vanlig
akrylamidgel er 250  l
APS og 25
l
APS og 25  l
TEMED. Bland godt før løsningen suges opp i en 60 ml sprøyte.
l
TEMED. Bland godt før løsningen suges opp i en 60 ml sprøyte.
- 7.
- Før gelløsningen forsiktig inn mellom glassplatene nedenfra mens det
bankes forsiktig på den øverste glassplaten der hvor gelfronten beveger seg.
Unngå bobler. Det er særdeles viktig å unngå bobler i lysveien til laseren.
Sett en klemme over glassplatene ved kammen. La gelen polymerisere minst en
time.
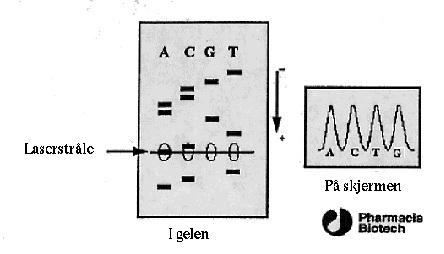
Figure 3.6: Prinsippet for ALFExpress sekvensering.
Cy 5 merkede DNA-fragmenter detekteres av fotoceller bak gelen når de når
laserstrålens lysvei. Signalene fra fotocellene plottes som funksjon av tiden.
Sekvens bestemmes ut fra rekkefølgen på toppene
|
Nedenfor er montering av gel, igangsettelse av elektroforese og datalogging
for ALFExpress beskrevet. Prosedyren gjelder ved bruk av
programvare-pakken ALFWin for MS Windows 95. Prinsippet for ALFExpress er
utdypet i figur 3.6.
- 1.
- Heng opp gelformen med polymerisert gel i ALFExpress og kobl til
forsyningsslangene for temperatur-regultert vann.
- 2.
- Sett i det nedre bufferkaret og fyll i 0,6xTBE (se avsnitt 3.3.1.3.5)
buffer i øvre og nedre kar.
- 3.
- Logg inn på PCen som brukes til å styre ALFExpress og lag din egen
bruker i programmet ALFWin Admin hvis du ikke har fra før. Dette for å
få resultater og loggdata til å lagres i et personlig område på disken.
- 4.
- Start programmet ALFWin Control. Åpne her en såkalt
casebook, som inneholder eksperimentelle betingelser. Man kan velge
mellom ulike varianter, X-longread er mye brukt dersom man ønsker å
lese langt. Den setter effekten til 25 W, temperatur i gelen til 55
 C,
elektroforesetiden til 15 timer og en del andre parametre. Endre på
betingelsene dersom det er ønskelig.
C,
elektroforesetiden til 15 timer og en del andre parametre. Endre på
betingelsene dersom det er ønskelig.
- 5.
- La programmet overføre de eksperimentelle paramtrene til ALFExpress
ved å klikke på knappen merket ``PRESET''. Vent til temperatur og laser er
justert (minst 15 minutter).
- 6.
- Appliser 3,5
 l
av hver sekvenserings-reaksjon (denaturert på forhånd), fortrinnsvis i
rekkefølgen ACGT for hvert templat. Dersom annen rekkefølge av basene benyttes
må dette spesifiseres i programvaren. Følg oppmerkingen på apparatet.
l
av hver sekvenserings-reaksjon (denaturert på forhånd), fortrinnsvis i
rekkefølgen ACGT for hvert templat. Dersom annen rekkefølge av basene benyttes
må dette spesifiseres i programvaren. Følg oppmerkingen på apparatet.
- 7.
- Monter elektroder og start elektroforesen ved å klippe på knapp merket
``START''.
- 8.
- Informasjon om prøver, spor og lagring av resultater kan lagres mens
kjøringen pågår. Velg normal prosessering som en såkalt ``post-run action''.
- 1.
- Start programmet ALFWin Evaluation
- 2.
- Åpne filen hvor resultatene ble lagret
- 3.
- Velg prosessering dersom dette ikke ble gjort automatisk etter kjøring.
Normalprosessering brukes i de aller fleste tilfeller. Ved prosesseringen
leser programvaren basesekvens ut av fluorescens-plottet. ALFEvalutation
tolker rekkefølgen toppene i fluorescens-plottet som rekkefølgen av baser.
- 4.
- Programvaren kan så benyttes til å korrekturlese og redigere den
prosesserte sekvensen.
Løsninger
Nedenfor er innholdet i de kommersielle løsningene levert med ``Thermo
Sequenase fluorescent labelled primer cycle sequencing kit'' (RPN 2436) fra
AMERSHAM gjengitt.
Tris-HCl (pH 9,5)
MgCl2
Tween 20
Nonidet P-40
2-mercaptoetanol
dATP, dCTP, dGTP, dTTP
ddATP
Termostabil pyrofosfatase
Thermo Sequenase DNA polymerase
Tris-HCl (pH 9,5), MgCl2, Tween 20, Nonidet P-40 ,
2-mercaptoetanol, dATP, dCTP, dGTP, dTTP, ddCTP, Termostabil pyrofosfatase,
Thermo Sequenase DNA polymerase
Tris-HCl (pH 9,5), MgCl2, Tween 20, Nonidet P-40 ,
2-mercaptoetanol, dATP, dCTP, dGTP, dTTP, ddGTP, Termostabil pyrofosfatase,
Thermo Sequenase DNA polymerase
Tris-HCl (pH 9,5), MgCl2, Tween 20, Nonidet P-40 ,
2-mercaptoetanol, dATP, dCTP, dGTP, dTTP, ddTTP, Termostabil pyrofosfatase,
Thermo Sequenase DNA polymerase
Formamid
EDTA
Metylfiolett
I det følgende er oppskriften på gelløsningene og bufferne anbefalt til
ALFExpress gjengitt.
37,5 ml 40% Akrylamid
25 ml 10xTBE (se
nedenfor)
(se
nedenfor)
105g urea
H2O til 250 ml
Lagres i kjøleskap på
lystett flaske
30 ml 50% Long Ranger Akrylamid
30 ml 10xTBE (se nedenfor)
(se nedenfor)
105g urea
H2O til 250 ml
Lagres i kjøleskap på lystett flaske
Merk at denne oppskriften er forskjellig fra vanlig 10xTBE (beskrevet i
tillegg A).
121,14 g Tris base
51,32 g borsyre
3,72 g EDTA
Filtreres gjennom
0,45 m filter for å unngå utfelling
m filter for å unngå utfelling
10 ml 100% etanol
15  l
iseddik
l
iseddik
2  l
Bind-Silan
l
Bind-Silan
Dette avsnittet behandler bruk av dataprogrammer til analyse av DNA-sekvens.
Da disse metodene er av en annen karakter enn de øvrige i oppgaven, har jeg
valgt å inndele beskrivelsen etter et noe annet mønster. Hver programgruppe
beskrives for seg, med en forenklet prinsipiell innledning og siden en
kortfattet omtale av bruken.
Søk etter homologe DNA-sekvenser med
fastA og blast
Programmene FastA og blast søker i databaser etter sekvenser
som er homologe til en oppgitt sekvens (query). FastA søker i
DNA-databaser, mens blast også kan søke i protein-databaser.
FastA søker etter likhet mellom en sekvens (query) og en gruppe
sekvenser (database-sekvenser) ved hjelp av metoden til PEARSON
& LIPMAN (1988) [110].
Først lager programmet en ``ordliste'' som inneholder alle mulige ``ord'' i
query-sekvensen. Med ord forstår vi alle nukleotidsekvenser med lengde n, som
settes av bruker. Posisjonen(e) hvor hvert ord forekommer i query-sekvensen
lagres i ordlisten.
Deretter søker programmet gjennom alle DNA-sekvensene i de ønskede databasene
og sammenligner hver enkelt med query-sekvensen. Hver sammenligning utføres ved
at det søkes etter likhet med ordene i ordlisten. Når likhet med et ord påvises,
gis det en score-verdi til sekvensen det sammenlignes med (normal score-verdi er
lik for alle ord).
Etter sammenligning summeres alle de enkelte score-verdiene for en sekvens
sammen. Sekvensene som gav høyest score, evalueres videre med en score-matrise
som tillater konserverte utbytninger og identitet i sekvenser kortere enn
ordlengden (< n). Total score for hver sekvens her lagres som init1.
Deretter undersøker programmet om noen av områdene med likheter i samme
sekvens ligger inntil hverandre. En ny parameter, initn beregnes som et
mål på størrelsen av de identiske strekkene. Til sist sammenstilles (alignes)
query-sekvensen med de beste sekvensene så langt ved hjelp av prosedyren til
CHAO, PEARSON og MILLER (1992) [111].
Sistnevnte prosedyre beregner også en parameter opt.
Programmet presenterer resultatet for bruker først ved et dotplot som viser
query-sekvensen nedover og antall treff (likhet med ord i ordlisten) som
funksjon av denne. Deretter listes sekvensene med høyest initn-verdi i
rekkefølge sammen med sine respektive init1, initn og
opt-paramaterne. Til sist vises sammenstillinger (alignments) av
query-sekvensene med de beste funnene.
Med søkeprogrammet blast (basic local alignment search tool)
kan query-sekvensen være enten DNA eller peptid. Det samme gjelder for
databasen. Blast kan også søke i peptid-databaser med DNA-query-sekvens
og motsatt. Da translaterer programmet DNA-sekvensen til peptid i alle faser før
homologi-søk.
Algoritmen til blast er basert på ALTSCHUL ET AL.
(1990) [112] og
opererer med et begrep som kalles MSP (Maximal-scoring Segment Pairs). Et
segment-par (SP) er et strekk med likhet mellom query-sekvensen og sekvensen det
sammenlignes med. Blast søker å utvide segmentparene helt til likheten
tar slutt. Deretter beregnes en score-verdi for segmentparet. Alle segmentpar
som er store nok til at de ikke forekommer tilfeldig med en relativ frekvens
større enn en forventningsverdi (expectation value), listes etter score-verdi.
Blast bruker mye kortere tid på homologi-søk enn fastA, men er
mindre sensitiv. FastA er bedre i stand til å detektere homologi på tross
av innsetninger, utbytninger og strekk uten idenitet internt i homologe
sekvenser.
FastA og blast er en del av gcg-pakken. Denne er tilgjengelig
blant annet på serveren bioslave ved Bioteknologisenteret i Oslo. Alle
kommandoer som gis i operativsystemet UNIX er her og i det følgende representert
ved symbolet > og så selve kommandoen med annerledes font. Symbolet >
representerer kommando-promptet og er ikke en del av kommadoen. For å oversende
kommandoen til kommandotolker (shell) må return-tasten benyttes. Hjelp for alle
kommandoene i gcg-pakken kan man få ved å gi kommandoen >
genhelp [navn på kommando].
- Fasta startest ved å gi kommandoen
> fasta -batch. Batch-opsjonen gis for å la programmet
gå i bakgrunnen. Da slipper man å vente mens programmet kjører (opptil flere
timer). Programmet ber så om en del parametre:
- Oppgi filnavn på query-sekvensen (sekvensen man vil undersøke for
homologi med database-sekvenser) når programmet. Query-sekvensen må være en
gcg-fil. Resultatet av homologisøket vil bli skrevet til fil med samme navn
som query-filen, men med ekstensjonen ``.fasta'' istedenfor ``.gcg''.
- Oppgi hvilken del av query-sekvensen som skal brukes til homologi-søk.
- Oppgi database(r) hvor det skal søkes for homologe sekvenser (f.eks.
GeneEMBL, Genebank og EMBL).
- Blast startes ved kommandoen >blast -batch. Oppgi
deretter på forespørsel følgende parametre:
- Oppgi query-sekvensens filnavn (gcg-fil). Blast gir ikke muligheten til
å velge ut bestemte deler av query-sekvensen.
- Oppgi databasens navn. Her gir programmet deg en liste med forslag. Velg
nummeret for den ønskede databasen eller kombinasjon av databaser.
- Oppgi statistisk forventningsverdi (normalverdi=10). Treff som
statistisk forventes å forekomme oftere enn denne verdien, ignoreres.
- Oppgi filnavn for resultat-utskriften.
Sammenligning av sekvenser med
compare og dotplot
Compare er et program som brukes til å sammenligne to sekvenser med
hensyn på homologi. Det søkes for homologi ved at et ``vindu'' på et visst
antall baser (oppgis av bruker) flyttes langs begge sekvensene. Programmet kan
finne punkter med identitet enten ved å detektere vinduer i begge sekvenser som
har et antall like nukleotider over en viss grenseverdi eller ved å detektere
helt identiske vinduer i begge sekvenser. Sistnevnte variant er 1000 ganger
raskere enn førstnevnte. Merk at vinduet flyttes gjennom hele den ene sekvensen
for hver vindu-posisjon i den andre sekvensen.
Resultatet av sammenligningen skrives til en fil som kan brukes til å lage et
todimensjonalt plot med programmet dotplot. De to sekvensene plottes på
hver sin akse. Forekomster av identitet mellom sekvensene vises ved et punkt i
koordinatsystemet. For eksempel vil identitet mellom vinduet som starter i
posisjon 20 i den ene x-sekvensen og i posisjon 350 i y-sekvensen, symboliseres
ved punktet (20,350). Fullstendig identiske sekvenser resulterer i en rett linje
diagonalt i koordinatsystemet.
Filer kan kopieres fra databaser og over til ditt filområde ved hjelp av
kommandoen > fetch [filnavn]. Filene som skal sammenlignes bør
ligge i samme katalog (i filsystemet), her bør man også være plassert ved start
av programmene.
- Start compare ved kommandoen > compare. Når programmet
ber om det, oppgi navnet på de to filene som skal sammenlignes. Oppgi også
størrelsen på vinduet. ( se avsnittet ovenfor) og navnet på resultatfilen.
- Skriv ut et dotplot med kommandoen > dotplot [filnavn],
hvor filnavn er navnet på en resultatfil laget av compare. Sørg for at
riktig printer er valgt i gcg-pakken før utskrift.
Programmet DNA Strider er et program for enkle analyser av DNA-sekvens
med grafisk brukergrensesnitt. Visning av seter for restriksjonsenzymer,
hypotetiske fordøyninger, lokalisering av åpne leserammer, translasjon og
aminosyrer er noen av mulighetene som foreligger. Dette avsnittet berører bare
de deler av programvaren som har vært brukt i min oppgave og er ikke tenkt å
være noen slags form for fullstendig brukermanual.
DNA Strider finnes foreløpig kun kompilert for MacOS, men kan kjøres
fra andre operativsystemer dersom Mac-emulatorer er installert. Programmet
startes ved å klippe på ikonet for programmet.
- Åpne DNA-sekvensen som skal analyseres. Dersom dette ikke er en
Strider-fil, men kun en tekstfil (ASCII) , kan sekvensen importeres.
Åpning og importering velges under menyen file.
- Under menyen enzymes kan man velge mellom mange alternativer, så
som forskjellige typer restriksjonskart og hypotetisk fordøyning.
- Under menyen translation kan man få frem kart over åpne leserammer
i alle faser, translatere DNA-sekvens og studere peptider med hensyn på
aminosyre-sammensetning.
![next]()
![up]()
![previous]()
![contents]()
Next: Diverse metoder Up: Metoder Previous: Kloning i Saccharomyces cerevisiae
Oddmund Nordgård
2001-07-17  til ALFExpress3.7
til ALFExpress3.7

